Why reduce the feature set?
결과적으로 모델의 복잡성을 줄이기 위해서이다. Feature가 많아질수록 모델이 많은 변수를 통해 작동하게된다. Neural Network의 구조를 살펴보자. 입력 레이어(Feature) - 히든 레이어 - 출력 레이어가 있다. 교육을 통해 혼련 데이터에서 가중치를 조정하여 네트워크가 모든 입력에 대해 최상의 가중치를 학습할 수 있다.
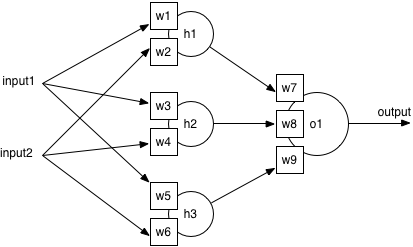
Feature가 많을수록 입력이 많아지므로 히든 레이어가 더 많은 가중치를 가지게 된다. 가중치를 조절하는데 많은 시간이 걸리므로 모델이 복잡할수록 교육 시간이 늘어난다는 의미이다. 다른 이유로는 작은 세트가 종종 더 정확한 모델로 이어진다는 사실이다. 이를 curse of dimensionality라고 한다. Feature가 많을수록 노이즈나 가짜 관계를 가지고 있을 확률이 높고 정확한 모델을 찾기 어려워질 수 있다는 것이다. 정확한 모델을 찾는데에 충분한 정보가 포함된 최적의 세트가 있을 것이며 중복 정보는 제거하거나 최소화 하는 방법을 찾을 것이다.
Feature selection
상관관계와 상호정보를 사용하여 Feature를 선택하고, 중복을 제거해본다.
Labelling the data
첫번째로 이전에 사용한 데이터 세트들을 불러온다. 데이터가 없을 경우 위의 코드를 실행한다.
basedir <- "../bearing_IMS/1st_test/"
library(e1071)
# Helper functions
fft.spectrum <- function (d)
{
fft.data <- fft(d)
# Ignore the 2nd half, which are complex conjugates of the 1st half,
# and calculate the Mod (magnitude of each complex number)
return (Mod(fft.data[1:(length(fft.data)/2)]))
}
freq2index <- function(freq)
{
step <- 10000/10240 # 10kHz over 10240 bins
return (floor(freq/step))
}
# Bearing data
Bd <- 0.331 # ball diameter, in inches
Pd <- 2.815 # pitch diameter, in inches
Nb <- 16 # number of rolling elements
a <- 15.17*pi/180 # contact angle, in radians
s <- 2000/60 # rotational frequency, in Hz
ratio <- Bd/Pd * cos(a)
ftf <- s/2 * (1 - ratio)
bpfi <- Nb/2 * s * (1 + ratio)
bpfo <- Nb/2 * s * (1 - ratio)
bsf <- Pd/Bd * s/2 * (1 - ratio**2)
all.features <- function(d)
{
# Statistical features
features <- c(quantile(d, names=FALSE), mean(d), sd(d), skewness(d), kurtosis(d))
# RMS
features <- append(features, sqrt(mean(d**2)))
# Key frequencies
fft.amps <- fft.spectrum(d)
features <- append(features, fft.amps[freq2index(ftf)])
features <- append(features, fft.amps[freq2index(bpfi)])
features <- append(features, fft.amps[freq2index(bpfo)])
features <- append(features, fft.amps[freq2index(bsf)])
# Strongest frequencies
n <- 5
frequencies <- seq(0, 10000, length.out=length(fft.amps))
sorted <- sort.int(fft.amps, decreasing=TRUE, index.return=TRUE)
top.ind <- sorted$ix[1:n] # indexes of the largest n components
features <- append(features, frequencies[top.ind]) # convert indexes to frequencies
# Power in frequency bands
vhf <- freq2index(6000):length(fft.amps) # 6kHz plus
hf <- freq2index(2600):(freq2index(6000)-1) # 2.6kHz to 6kHz
mf <- freq2index(1250):(freq2index(2600)-1) # 1.25kHz to 2.6kHz
lf <- 0:(freq2index(1250)-1) # forcing frequency band
powers <- c(sum(fft.amps[vhf]), sum(fft.amps[hf]), sum(fft.amps[mf]), sum(fft.amps[lf]))
features <- append(features, powers)
return(features)
}
# Set up storage for bearing-grouped data
b1m <- matrix(nrow=0, ncol=(2*23))
b2m <- matrix(nrow=0, ncol=(2*23))
b3m <- matrix(nrow=0, ncol=(2*23))
b4m <- matrix(nrow=0, ncol=(2*23))
# and for timestamps
timestamp <- vector()
for (filename in list.files(basedir))
{
cat("Processing file ", filename, "\n")
ts <- as.character(strptime(filename, format="%Y.%m.%d.%H.%M.%S"))
data <- read.table(paste0(basedir, filename), header=FALSE, sep="\t")
colnames(data) <- c("b1.x", "b1.y", "b2.x", "b2.y", "b3.x", "b3.y", "b4.x", "b4.y")
# Bind the new rows to the bearing matrices
b1m <- rbind(b1m, c(all.features(data$b1.x), all.features(data$b1.y)))
b2m <- rbind(b2m, c(all.features(data$b2.x), all.features(data$b2.y)))
b3m <- rbind(b3m, c(all.features(data$b3.x), all.features(data$b3.y)))
b4m <- rbind(b4m, c(all.features(data$b4.x), all.features(data$b4.y)))
timestamp <- c(timestamp, ts)
}
cnames <- c("Min.x", "Qu.1.x", "Median.x", "Qu.3.x", "Max.x", "Mean.x", "SD.x", "Skew.x", "Kurt.x", "RMS.x", "FTF.x", "BPFI.x", "BPFO.x", "BSF.x", "F1.x", "F2.x", "F3.x", "F4.x", "F5.x", "VHF.pow.x", "HF.pow.x", "MF.pow.x", "LF.pow.x", "Min.y", "Qu.1.y", "Median.y", "Qu.3.y", "Max.y", "Mean.y", "SD.y", "Skew.y", "Kurt.y", "RMS.y", "FTF.y", "BPFI.y", "BPFO.y", "BSF.y", "F1.y", "F2.y", "F3.y", "F4.y", "F5.y", "VHF.pow.y", "HF.pow.y", "MF.pow.y", "LF.pow.y")
colnames(b1m) <- cnames
colnames(b2m) <- cnames
colnames(b3m) <- cnames
colnames(b4m) <- cnames
b1 <- data.frame(timestamp, b1m)
b2 <- data.frame(timestamp, b2m)
b3 <- data.frame(timestamp, b3m)
b4 <- data.frame(timestamp, b4m)
write.table(b1, file=paste0(basedir, "../b1_all.csv"), sep=",", row.names=FALSE)
write.table(b2, file=paste0(basedir, "../b2_all.csv"), sep=",", row.names=FALSE)
write.table(b3, file=paste0(basedir, "../b3_all.csv"), sep=",", row.names=FALSE)
write.table(b4, file=paste0(basedir, "../b4_all.csv"), sep=",", row.names=FALSE)이후 파일을 읽어준다.
b1 <- read.table(file=paste0(basedir, "../b1_all.csv"), sep=",", header=TRUE)
b2 <- read.table(file=paste0(basedir, "../b2_all.csv"), sep=",", header=TRUE)
b3 <- read.table(file=paste0(basedir, "../b3_all.csv"), sep=",", header=TRUE)
b4 <- read.table(file=paste0(basedir, "../b4_all.csv"), sep=",", header=TRUE)이후 데이터에 class label을 추가해주어야 한다. 베어링 테스트에 대한 결과에 따르면 베어링 3번은 내부 오류로 인해 실패하였으며 베어링 4번은 롤링 요소로 실패하였다고 나타난다. 하지만 베어링 1번도 수상하며 베어링 2번도 문제가 생기기 시작했다는 것을 알 수 있다. 라벨은 다음과 같다.
early : green
normal : blue
suspect : yellow
failure : red
unknown : black
라벨 없는 그래프를 그려보자.
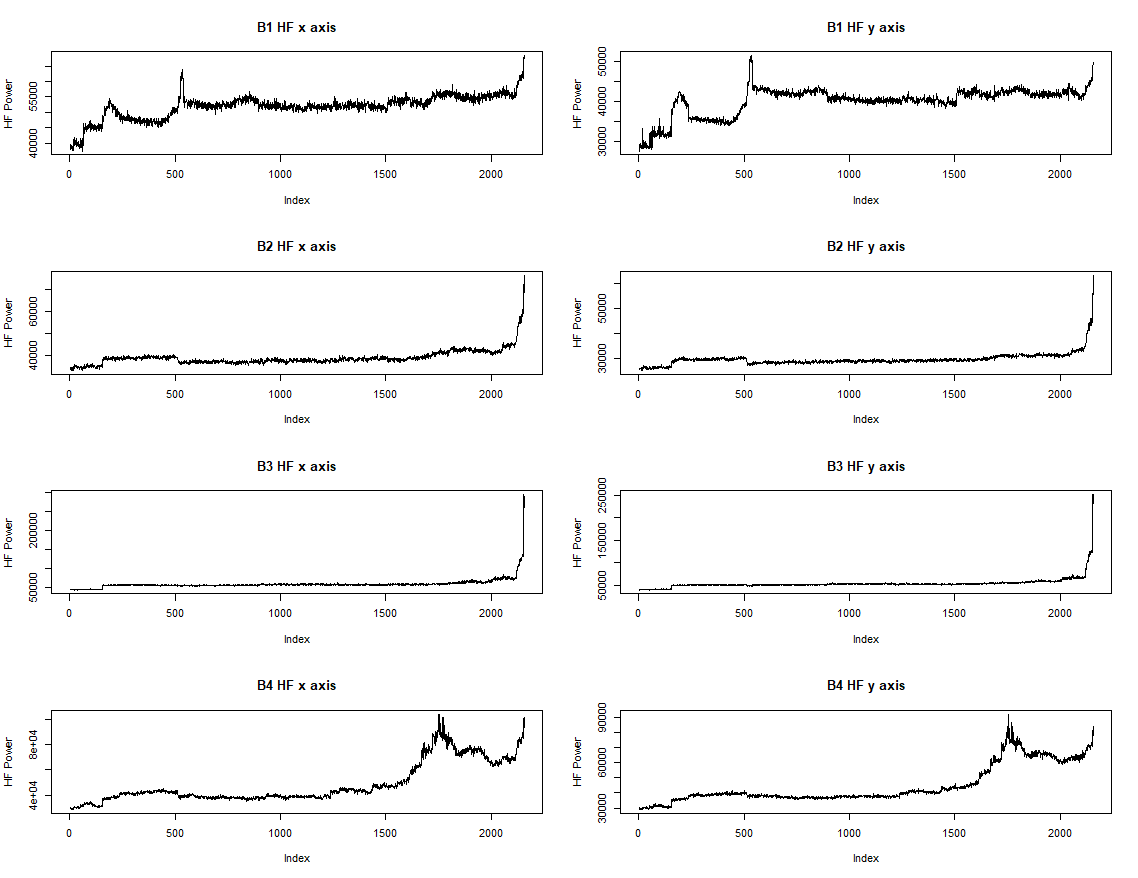
다 아는 데이터로 라벨을 추가해서 다시 그려본다.
Correlation
feature 선택의 첫 단계는 feature 사이의 상관관계를 알아내는 것이다. 상관관계는 -1과 1 사이의 값으로 정량화 할 수 있는데 0은 상관 없음, 1이면 선형 상관관계, -1은 반비례 한다는 것을 의미한다. 지금의 경우 높은 상관 관계를 가지고 있는 feature는 제거하는 것이 좋다. 동일한 정보는 포함하지 않는 것이 차원 축소에 이득이기 때문이다. 또한 상관 관계를 찾아낼 때에는 'normal' 상태일 때의 데이터를 사용하는 것이 좋다. 이는 베어링이 정상 작동을 하고 있지 않을 때에 어떤 변수가 영향을 받는지 모르기 때문이다.
상관관계를 분석하지 않아도 되는 데이터는 가장 큰 FFT 요소, 상태 레이블, 타임 스탬프 등이 있다. 이를 고려하여 데이터셋을 구성한다.
norm <- rbind(
cbind((b1[(b1$State == "normal"),-c(1, 16, 39, 48)]), bearing="b1"),
cbind((b2[(b2$State == "normal"),-c(1, 16, 39, 48)]), bearing="b2"),
cbind((b3[(b3$State == "normal"),-c(1, 16, 39, 48)]), bearing="b3"),
cbind((b4[(b4$State == "normal"),-c(1, 16, 39, 48)]), bearing="b4")
)이후 상관관계를 나타내기 위해 library를 불러와준다.
install.packages("corrgram")
library(corrgram)
corrgram(norm)
install.packages("corrplot")
library(corrplot)파란색은 양의 상관관계이며 빨간색은 음의 상관관계이다. 흰색과 옅은 색은 상관 관계가 거의 없거나 전혀 없음을 의미한다.
cor <- cov2cor(cov(norm[, -45]))
alikes <- apply(cor, 2, function(col) { names(Filter(function (val) { val > 0.9 }, sort.int(abs(col), decreasing=TRUE))) } )
cat(str(alikes, vec.len=10))
corrplot(cor,method="square", tl.pos="n")
0.9이상 혹은 0.9이하의 상관관계를 가지고 있는 친구들을 나열해보자.
List of 44
$ Min.x : chr "Min.x"
$ Qu.1.x : chr [1:5] "Qu.1.x" "RMS.x" "LF.pow.x" "SD.x" "Qu.3.x"
$ Median.x : chr "Median.x"
$ Qu.3.x : chr [1:5] "Qu.3.x" "SD.x" "RMS.x" "LF.pow.y" "Qu.1.x"
$ Max.x : chr "Max.x"
$ Mean.x : chr "Mean.x"
$ SD.x : chr [1:5] "SD.x" "RMS.x" "Qu.3.x" "Qu.1.x" "LF.pow.y"
$ Skew.x : chr "Skew.x"
$ Kurt.x : chr "Kurt.x"
$ RMS.x : chr [1:4] "RMS.x" "SD.x" "Qu.1.x" "Qu.3.x"
$ FTF.x : chr "FTF.x"
$ BPFI.x : chr "BPFI.x"
$ BPFO.x : chr "BPFO.x"
$ BSF.x : chr "BSF.x"
$ F2.x : chr "F2.x"
$ F3.x : chr "F3.x"
$ F4.x : chr "F4.x"
$ F5.x : chr "F5.x"
$ VHF.pow.x: chr [1:2] "VHF.pow.x" "VHF.pow.y"
$ HF.pow.x : chr [1:4] "HF.pow.x" "MF.pow.x" "HF.pow.y" "SD.y"
$ MF.pow.x : chr [1:7] "MF.pow.x" "HF.pow.y" "SD.y" "HF.pow.x" "RMS.y" "Qu.3.y" "VHF.pow.y"
$ LF.pow.x : chr [1:2] "LF.pow.x" "Qu.1.x"
$ Min.y : chr "Min.y"
$ Qu.1.y : chr [1:4] "Qu.1.y" "RMS.y" "LF.pow.y" "SD.y"
$ Median.y : chr [1:2] "Median.y" "Mean.y"
$ Qu.3.y : chr [1:6] "Qu.3.y" "SD.y" "VHF.pow.y" "MF.pow.x" "HF.pow.y" "LF.pow.y"
$ Max.y : chr "Max.y"
$ Mean.y : chr [1:2] "Mean.y" "Median.y"
$ SD.y : chr [1:9] "SD.y" "VHF.pow.y" "MF.pow.x" "RMS.y" "HF.pow.y" "Qu.3.y" "LF.pow.y" "Qu.1.y" "HF.pow.x"
$ Skew.y : chr "Skew.y"
$ Kurt.y : chr "Kurt.y"
$ RMS.y : chr [1:7] "RMS.y" "Qu.1.y" "SD.y" "LF.pow.y" "VHF.pow.y" "MF.pow.x" "HF.pow.y"
$ FTF.y : chr "FTF.y"
$ BPFI.y : chr "BPFI.y"
$ BPFO.y : chr "BPFO.y"
$ BSF.y : chr "BSF.y"
$ F2.y : chr "F2.y"
$ F3.y : chr "F3.y"
$ F4.y : chr "F4.y"
$ F5.y : chr "F5.y"
$ VHF.pow.y: chr [1:8] "VHF.pow.y" "SD.y" "Qu.3.y" "RMS.y" "VHF.pow.x" "MF.pow.x" "LF.pow.y" "HF.pow.y"
$ HF.pow.y : chr [1:8] "HF.pow.y" "MF.pow.x" "SD.y" "MF.pow.y" "HF.pow.x" "VHF.pow.y" "Qu.3.y" "RMS.y"
$ MF.pow.y : chr [1:2] "MF.pow.y" "HF.pow.y"
$ LF.pow.y : chr [1:8] "LF.pow.y" "Qu.1.y" "RMS.y" "SD.y" "SD.x" "VHF.pow.y" "Qu.3.x" "Qu.3.y"하지만 이 정보로 feature를 뽑아내는 것은 쉽지 않다. 이전의 정보를 활용해 베스트를 뽑아낸다.
best <- c("Min.x", "Median.x", "Max.x", "Mean.x", "Skew.x", "Kurt.x", "FTF.x", "BPFI.x", "BPFO.x", "BSF.x", "F2.x", "F3.x", "F4.x", "F5.x", "Min.y", "Max.y", "Skew.y", "Kurt.y", "FTF.y", "BPFI.y", "BPFO.y", "BSF.y", "F2.y", "F3.y", "F4.y", "F5.y", "Qu.1.x", "VHF.pow.x", "Qu.1.y", "Median.y", "HF.pow.y")best라는 state를 추가하여 데이터를 생성한다.
data <- rbind(
cbind(bearing="b1", (b1[,c("State", best)])),
cbind(bearing="b2", (b2[,c("State", best)])),
cbind(bearing="b3", (b3[,c("State", best)])),
cbind(bearing="b4", (b4[,c("State", best)]))
)
write.table(data, file=paste0(basedir, "../all_bearings.csv"), sep=",", row.names=FALSE)Mutal information
베어링의 상태를 분류하는 가장 중요한 기능을 찾아내야한다. 정보는 베어링 상태의 독립 및 조건부 엔트로피의 차이로 계산된다. 상호정보는 entropy 패키지를 이용해 다음과 같이 계산할 수 있다.
> library("entropy")
> H.x <- entropy(table(data$State))
> mi <- apply(data[, -c(1, 2)], 2, function(col) { H.x + entropy(table(col)) - entropy(table(data$State, col))})
> sort(mi, decreasing=TRUE)
Skew.x Kurt.x FTF.x BPFI.x BPFO.x BSF.x
1.4318662 1.4318662 1.4318662 1.4318662 1.4318662 1.4318662
Skew.y Kurt.y FTF.y BPFI.y BPFO.y BSF.y
1.4318662 1.4318662 1.4318662 1.4318662 1.4318662 1.4318662
VHF.pow.x HF.pow.y Mean.x Qu.1.x Qu.1.y F5.y
1.4318662 1.4318662 1.4026494 0.5539613 0.5328388 0.5225875
F5.x F4.y F3.y F4.x F2.y F3.x
0.5007170 0.4942429 0.4937412 0.4800929 0.4558328 0.4472345
Max.x Min.x Max.y Min.y F2.x Median.y
0.3743911 0.3487148 0.3460297 0.3381148 0.2632983 0.2313810
Median.x
0.2261523흥미롭게도 처음의 14가지 features는 모두 베어링 상태에 대해 동일하다. 목록의 맨 위 기능을 사용하고 아래 기능은 제거해준다. 이 때 RPART라는 의사결정트리 분류자를 사용한다.
library("rpart")
library("caret")
train.rows <- seq(1, length(data$State), by=4) # every fourth index
sorted <- names(sort(mi, decreasing=TRUE))
accuracies <- vector()
for (i in 2:length(sorted))
{
form <- paste0("data$State ~ data$", Reduce(function (r, name) { paste(r, paste0("data$", name), sep=" + ") }, sorted[1:i]))
model <- rpart(as.formula(form), data, subset=train.rows)
pred <- predict(model, data, type="class") # contains train and test set predictions
cm <- confusionMatrix(pred[-train.rows], data[-train.rows, 2]) # only the non-training rows
# pull out the answer
accuracies <- c(accuracies, cm["overall"][[1]]["Accuracy"][[1]])
}
plot(accuracies, xlab="Number of features", ylab="Accuracy")
Conclusion
46가지 feature를 14가지로 줄여보았다. 이 때에 결과가 덜 복잡하고 빠르게 교육하면 전체 기능을 사용하는 것보다 정확해지는 경향을 확인할 수 있었다.
'미분류 > R' 카테고리의 다른 글
| Understanding data science: classification with neural networks in R (0) | 2019.06.12 |
|---|---|
| Understanding data science: clustering with k-means in R (0) | 2019.06.11 |
| R 기본 (추가 중) (0) | 2019.06.05 |
| Understanding data science:designing useful features with R (0) | 2019.06.03 |
| Understanding data science: feature extraction with R (0) | 2019.06.03 |



